种质研究室围绕农业供给侧结构性改革以及食物、健康、资源环境安全和农民增收等重大战略需求和关键难题,开展油料种质资源的收集、评价鉴定和保护、新基因鉴定与发掘利用、优异新种质创新利用等课题研究,为育种和产业发展提供各类具有重大利用价值的优异新种质或新基因。承担国家重点研发计划项目、国家科技支撑、863计划、国家自然科学基金等重大课题30余项,发表论文110余篇、获成果奖励6项、专利授权4项、主编参编著作5部、招收培养博士后、博士和硕士研究生21人,组织建成具有世界先进水平的国家油料中期种质库和油料种质资源共享平台,创建了中国油菜种质资源基础和核心收集品,发掘、创制和利用一大批具有丰产、优质、抗病虫和养分高效的优异种质,有效支撑和促进了油料科研与产业的高效和可持续发展。
研究方向
1、油菜种质资源实物与信息共享平台建设
广泛收集国内外油菜及其野生近缘种种质资源,开展系统深入的评价鉴定,建立国家油料作物种质资源实物与信息平台,为油菜育种、基础研究和产业的可持续发展奠定坚实的物质基础。

2、油菜优异种质和新基因的发掘、鉴定与利用
综合利用现代生物科学和信息科学等最新技术手段,创建规模化油菜表型精准鉴定技术体系和高效的油菜全基因组基因型鉴定技术平台,依据育种新需求,规模化发掘和鉴定高产、优质、抗病虫、抗逆、养分高效和适合机械化等优异种质和新基因,为油菜育种新突破提供理论、技术和材料支撑。
3、优异油料作物种质资源的创新
研究油菜优异种质和重要性状形成机制,综合利用常规育种技术、远缘杂交、诱变和分子标记辅助选择等现代生物技术手段,建立创制新种质的新理论与新技术体系,利用野生种等开展新种质分子设计与创新,创制突破性种质,突破限制油菜育种和产业发展的限制性瓶颈。
4、特色油菜种质资源的开发和利用
依据农业供给侧结构性改革需求,研究油菜种质资源的用途多样性,探索广泛利用种质资源多样性的有效途径,通过发掘、创制和利用多彩油菜、特高硫苷等特色种质资源,打造特色油菜为核心的新产业,拓展油菜产业链和价值链,实现农民增收。

近期承担的主要课题
1、国家重点研发计划项目(National Key Program for Research and Development)
主要经济作物种质资源精准鉴定与创新利用 ( 2016YFD0100202 )
2、国家863和973课题 (National 863 Hi-tech and 973 Basic Research Projects)
1)油料作物优异亲本形成的遗传基础和优良基因资源合理组配与利用-油菜杂种品种优异亲本形成的遗传解析与应用(973)
2)油菜籽油脂形成的分子生物学机制及其代谢调控-油菜种质资源中高含油量等位基因的鉴定分析
3)油菜高油酸基因的克隆及功能分析-油菜高油酸基因的克隆及功能分析(973)
4)油菜品种分子设计的生物信息数据库建设 (863)
3、国家自然科学基金项目(National Natural Science Foundation of China)
1)基于重测序的全基因组关联分析发掘油菜种子耐热等位基因(31601341)
2)芸薹属油菜类作物细胞质基因组单倍型的精细解析及其协同进化分析(31600179)
3)全基因组关联分析鉴定控制油菜种子低积累重金属镉的关键基因(31470088)
4)利用全基因组SNP关联分析发掘甘蓝型油菜氮高效等位基因(31301360)
5)甘蓝型油菜叶绿体基因组特定区域高分辨率单倍型图谱的构建与分析(31100911)
6)DNA甲基化在油菜小孢子胚胎发生过程中的调控机制研究(31100236)
7)油菜花瓣缺失突变及其遗传操纵的分子机制研究(30170585)
4、国家科技攻关和科技支撑项目(National S&T Support Project)
1)油料作物种质资源发掘与创新利用(十二五国家科技支撑项目,2013BAD01B03-08)
2)农作物种质资源收集编目入库与安全保存(十二五国家科技支撑项目, 2013BAD01B0102)
3)农作物种子质量检验及加工技术研究与产业化(国家科技支撑项目2011BAD35B09)4)油料、糖料种质资源创新与利用研究(国家十五重点科技攻关项目2001BA511B07)
5)油料、糖料种质资源创新利用研究(国家十五重点科技攻关项目2004BA525B07)
5、其它国家和省部级资助项目(Other national and provincial projects)
1)国家农作物种质资源平台油料作物种质资源子平台(国家科技基础条件平台项目,NICGR2016-014)
2)油料作物种质资源收集、编目与繁种入库(农业部资源保护项目)
3)育种急需的甘蓝型油菜新种质的创制与利用(湖北省科技支撑计划项目,2015BBA189)
4)甘蓝型油菜叶绿体基因组特定区域高分辨率单倍型图谱的构建与分析(湖北省自然科学基金,2011CDB353 )
5)油菜高油酸品种的分子标记辅助选育(湖北省自然科学基金)
6、院所长基金(Director Foundation project)
1) 江西红壤野外观测站油菜种质资源的观测鉴定与共享数据库建设
2) 欧洲原始野生甘蓝重要性状评价鉴定与创新利用
3) 油菜基因资源的表型精准鉴定与细胞质基因组DNA多态性研究
4) 油菜核心资源的表型鉴定和基因型鉴定
7、国际合作项目(International cooperation project)
1) 国外油料作物种质资源引进与繁殖鉴定(国际科技合作与交流专项 2010DFB33340)
2) 国外优异和特色作物种质资源引进与利用(948项目,2011-G1-03)
3) 抗病和抗虫油菜的遗传改良(中国-丹麦国际合作项目)
主要科研成果
1. 省部级以上科技成果奖励
1) 中国农作物种质资源收集保存评价与利用---国家科技进步一等奖(集体)
2) 我国油菜种质资源搜集研究与利用---农业部科技进步二等奖
3) 新疆野生油菜种质资源的发现与研究---新疆维吾尔自治区科技进步二等奖
4) 贵州省油菜基因库的创建---贵州省科技进步三等奖
5) 陕西省油菜种质资源搜集评价利用研究---陕西省科技进步三等奖
6) 云南油菜种质资源的搜集繁种鉴定及特异种质研究---云南省科技进步三等奖
2. 已获国家专利
1) 一种广谱抗虫转基因油菜的培育方法(国家发明专利,CN200810046824.7)。
2) 具有高类胡萝卜素的油料作物的培育方法(国家发明专利,CN200710052841.7)。
3) 一种超声辅助农杆菌介导的植物in planta遗传转化方法(国家发明专利201010298516.0)。
4) 一种带蓄水池的高通量种子发芽盒(中国实用新型专利,201620588263.3)。
3. 代表性著作
1) 《主要作物起源起源与发展——油菜》2002,农业出版社,北京。
2) 《油菜种质资源数据标准和描述规范》,2007,中国农业出版社,北京。
3) 《中国农作物及其野生近缘植物,经济作物,第三章》,2007,中国农业出版社,北京。
4) 《中国油菜品种资源目录(续编一)》,1993,中国农业出版社,北京。
5) 《中国油菜品种资源目录(续编二)》,1997,中国农业科技出版社,北京。
4. 代表性论文
1) Li H, Zhang L, Hu J, Zhang F, Chen B, Xu K, Gao G, Li H, Zhang T, Li Z and Wu X Genome-Wide Association Mapping Reveals the Genetic Control Underlying Branch Angle in Rapeseed (Brassica napus L.). Frontier in plant science, 2017,8:1054. doi: 10.3389/fpls.2017.01054
2) Chen Biyun; Xu Kun; Li Hao; Gao Guizhen; Yan Guixin; Qiao Jiangwei; Wu Xiaoming, Evaluation of quality traits and their genetic variation in global collections of Brassica napus L, Plant Genetic Resources: Characterization and Utilization, 2017,1-10.
3) Wang LM, Jin X, Li QB, Wang XC, Li ZY, Wu XM*. Comparative Proteomics Reveals that Phosphorylation of Carbonic Anhydrase 1 Might be Important for Adaptation to Drought Stress in Brassica napus. Scientific Reports, 2016, SREP-16-25909B doi: 10.1038/srep39024
4) Yan GX, Lv XD, Gao GZ, Li F, Qiao JW, Li J, Xu K, Chen BY, Wang LM, Xiao X, Wu XM*. Identification and characterization of a glyoxalase I gene in a rapeseed cultivar with seed thermos tolerance. Frontier in plant science, 2016, 7: 150
5) Wang N, Chen BY, Xu K, Gao GZ, Li F, Qiao JW, Yan GX, Li J, Li H and Wu XM*. Association Mapping of Flowering Time QTLs and Insight into Their Contributions to Rapeseed Growth Habits. Frontier in plant science, 2016,7: 1-11
6) Li LX, Luo YJ, Chen BY, Xu K, Zhang FG, Li H, Huang Q, Xiao X, Zhang TY, Hu JH, Li F and Wu XM*. Genome-Wide Association Study Reveals New Loci for Resistance to Clubroot Disease in Brassica napus. Frontier in plant science, 2016, 7: 1483
7) Li J, Huang Q, Sun MX, Zhang TY, Li H, Chen BY, Xu K, Gao GZ, Li F, Yan GX, Qiao JW, Cai YP, Wu XM*. Global DNA methylation variations after short-term heat shock treatment in cultured microspores of Brassica napus cv. Topas. Scientific Reports, 2016, 6:38401 DOI: 10.1038/srep38401
8) Li F, Chen BY, Xu K, Gao GZ, Yan GX, Qiao JW, Li J, Li H, Li LX, Xiao X, Zhang TY, Nishio K, Wu XM*. A genome-wide association study of plant height and primary branch number in rapeseed (Brassica napus L.). Plant Science, 2016, 242:169-77.
9) Qiao JW, Cai MX, Yan GX, Wang N, Li F, Chen BY, Gao GZ, Xu K, Li J and Wu, XM*. High-throughput multiplex cpDNA resequencing clarifies the genetic diversity and genetic relationships among Brassica napus, Brassica rapa and Brassica oleracea. Plant Biotechnol. J., 2015, doi: 10.1111/pbi.12395
10) Yan GX, Li D, Cai MX, Gao GZ, Chen BY, Xu K, Li J, Li F, Wang N, Qiao JW, Li H, Zhang TY, Wu XM*. Characterization of FAE1 in the zero erucic acid germplasm of Brassica rapa L. Breeding Science, 2015, 65:1-8.
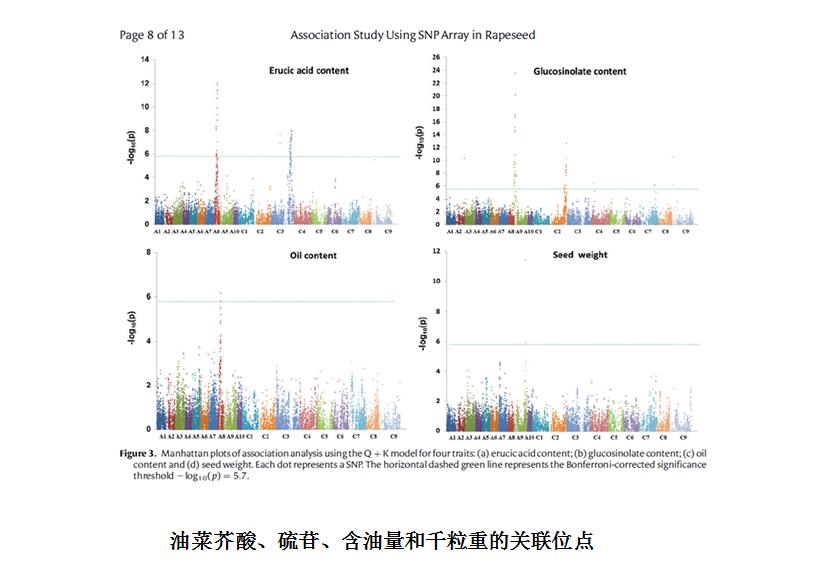
11) Li F, Chen BY; Xu K, Wu JF, Song WL, Bancroft I, Harper A, Trick M, Liu SY, Gao GZ, Wang N, Yan GX, Qiao JW, Li J, Li H, Xiao X, Zhang TY, Wu XM*, Genome-Wide Association Study Dissects the Genetic Architecture of Seed Weight and Seed Quality in Rapeseed (Brassica napus L.). DNA Research, 2014, 21:355-367.
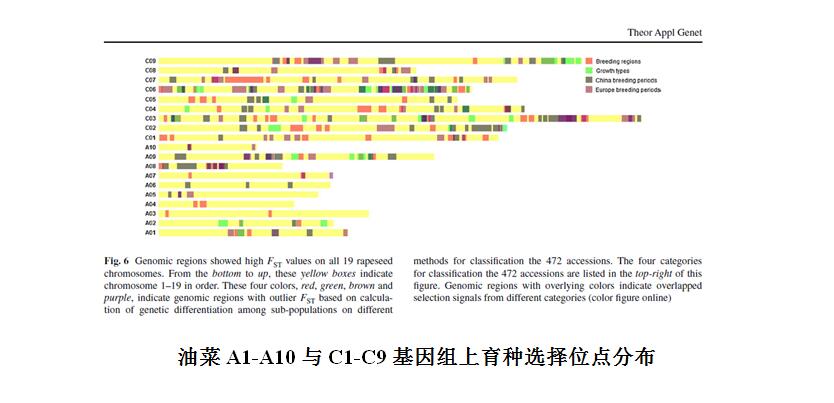
12) Wang N, Li F, Chen BY, Xu K, Yan GX, Qiao JW, Li J, Gao GZ, Bancroft I, Meng JL, King GJ., Wu XM*, Genome‑wide investigation of genetic changes during modern breeding of Brassica napus. Theor Appl Genet, 2014, 127:1817-1829.
13) Gao GZ, Li J, Li H, Li F, Xu K, Yan GX, Chen BY, Qiao JW and Wu XM*, Comparison of the heat stress induced variations in DNA methylation between heat-tolerant and heat-sensitive rapeseed seedlings, Breeding Science, 2014, 64: 125–133.
14) Wu JF, Li F, Xu K, Gao GZ, Chen BY, Yan GX, Wang N, Qiao JW, Li J, Li H, Zhang TY, Song WL and Wu XM* Assessing and broadening genetic diversity of a rapeseed germplasm collection, Breeding Science, 2014, 64: 321-330.
15) Chen BY, Xu K, Li J, Li F, Qiao JW, Li H, Gao GZ, Yan GX, Wu XM*, Evaluation of yield and agronomic traits and their genetic variation in 488 global collections of Brassica napus L., Genet Resour Crop Evol, 2014, 61:979-999.
16) Li J, Gao GZ, Xu K, Chen BY, Yan GX, Li F, Qiao JW , Zhang TY, Wu XM*, Genome-Wide Survey and Expression Analysis of the Putative Non-specific Lipid Transfer Proteins in Brassica rapa L., Plos One, 2014, 9(1): e84556. doi:10.1371/journal.pone.0084556
17) Li J, Gao GZ, Zhang TY, Wu XM*. The Putative Phytocyanin Genes in Chinese cabbage (Brassica rapa L.): Genome-wide Identification, Classification and Expression Analysis. Molecular Genetics and Genomics, 2013, 288: 1-20.
18) Zeng CL, Wang GY, Wang JB, Yan GX, Chen BY, Xu K, Li J, Gao GZ, Wu XM*, High-Throughput Discovery of Chloroplast and Mitochondrial DNA Polymorphisms in Brassicaceae Species by ORG-EcoTILLING, PLOS ONE, 2012, 7(11): e47284. doi:10.1371/journal.pone.0047284
19) Li J, Wu XM*. Genome-wide identification, classification and expression analysis of genes encoding putative fasciclin-like arabinogalactan proteins in Chinese cabbage (Brassica rapa L.). Molecular Biology Reports, 2012, 39: 10541-10555.
20) Yan GX, Wu XM*, Li Dan, Zeng CL, Lv PJ, Gao GZ, Chen BY, Xu K, Lv XD. AssessingHigh-Resolution melt curve analysis for accurate detection of DNA polymorphisms in the chloroplast gene accD of crucifer species. Biochemical Systematics and Ecology, 2012, 44: 352–360.


